
汲んだ水から魚を数える
-環境DNA分析による個体数の推定法を実証-
(筑波研究学園都市記者会、環境省記者クラブ、環境記者会、宮城県政記者会、島根県政記者会、京都大学記者クラブ、北海道教育庁記者クラブ、兵庫県教育委員会記者クラブ、神戸民間放送記者クラブ、大阪科学・大学記者クラブ同時配付)
| 令和2年7月3日(金) 国立環境研究所 東北大学 島根大学 京都大学 北海道大学 神戸大学 |
|
国立環境研究所 深谷肇一 特別研究員、東北大学 近藤倫生 教授、島根大学 南憲吏 助教、京都大学 山本哲史 助教、益田玲爾 教授、北海道大学 笠井亮秀 教授、宮下和士 教授、神戸大学 源利文 准教授らの共同研究チームは、水中に含まれる生物由来のDNA(環境DNA)の分析に基づいて対象の生物の個体数を推定する新手法を開発しました。さらに、京都府舞鶴湾全域に生息するマアジを対象に本手法を利用することで、海洋環境において環境DNAの濃度計測から生物の個体数を推定できることを初めて実証しました。目視や捕獲による調査を行うことなく、水試料の収集と分析のみによって水生生物の個体数を正確に把握できる可能性が示されたことで、海や湖などの水域生態系を定量的にモニタリングするための効率的な新技術の実現につながることが期待されます。 本研究の成果は、令和2年7月1日付で生態学分野の学術誌「Molecular Ecology」に電子版が掲載されました。 |
背景
自然環境中の水や土、空気などには、動物や植物の体から遊離した細胞や組織の断片が含まれています。近年の研究から、これら環境中の細胞・組織よりDNA(環境DNA)を抽出・分析すれば、生物の生息状況を把握できることが分かってきました。従来利用されてきた目視や捕獲による直接的な調査と比べて労力が少なくて済むことなどから、環境DNA分析は、生態系を効率的に観測するための新しい手法として注目を集めています。
環境DNA分析を用いると、環境中のDNAの有無を手がかりとして対象の生物が生息しているかどうかを判断できます。この方法は、対象の生物の分布(どこにいるのか)を調べる上で役立ちます。一方、生物の個体数が多ければ環境DNAも多く放出されると予想されるため、環境DNAの濃度を手がかりとすれば、生物の個体数(どれだけいるのか)も把握できるのではないかと期待されてきました。しかし、自然環境では、環境DNAは水の流れなどによって移動し、さらに時間が経てば分解してしまいます。そのため、一般的に、環境DNAの濃度と生物の個体数は単純な比例関係にはならず、環境DNA分析による個体数の推定は未だ解決できない困難な問題として残されていました。
内容
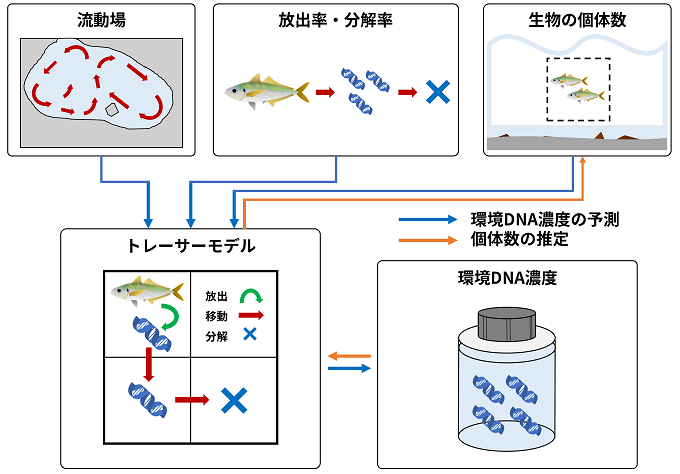
本研究では、環境DNA分析データから個体数を推定する上で特に考慮すべき要因として、(1)生物からのDNAの放出、(2)水中でのDNAの移動、および(3)DNAの分解に着目しました。環境DNAの濃度が高いからといって必ずしも個体数が多いとは言えないのは、これらの要因が複雑に作用するためです。環境DNA分析によって個体数を見積もるためには、こうした要因に左右される環境DNAの「一生」を踏まえることが不可欠だと考えられます。この考えに基づいて、私たちは「トレーサーモデル」と呼ばれる数理モデルを用いた、個体数推定の方法を開発しました(図1)。トレーサーモデルとは、対象の水域で物質(ここでは環境DNA)がどのように運ばれるかを表す物理モデルです。環境DNAのトレーサーモデルは、海洋力学シミュレーションなどから得られた流動場(※1)の情報と、生物の水槽飼育実験などから得られた環境DNAの放出率や分解率の値を組み合わせることで構築されます。トレーサーモデルを用いることで、対象水域のどこにどれくらいの数の生物が分布していれば、結果として、環境DNAがどのように分布するかを予測できます(図1、青の矢印)。しかし、私たちの目的は生物の分布から環境DNAの分布を予測することではなく、逆に環境DNAの分布から生物の分布を推定することです。そこで、私たちは、このモデルを「逆向きに解く(環境DNAの分布を手がかりに個体数の分布を求める)」ための統計手法を開発しました(図1、オレンジの矢印)。これによって初めて、対象の生物や水域に合わせたトレーサーモデルと、環境DNA濃度の多地点計測から、生物の個体数を見積もることが可能となりました。この手法は、河川や湖沼、海洋などの水域環境一般に適用することができます。
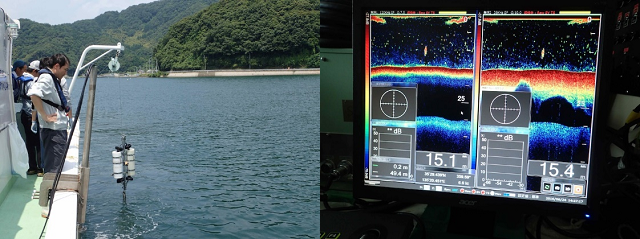
私たちは、2016年6月に京都府北部の舞鶴湾で野外調査を行い、マアジを対象に開発した手法を適用しました。この時期は体長3 cm程のマアジの幼魚(図2)が非常に多くの数を占める季節であることから、湾内のマアジの幼魚を数えることを目標としました。湾内の100地点を船で周り、表層・中層(海面から5m下)・底層(海底から1m上)からそれぞれ1リットルの海水を採集しました(図3左)。集めた海水を実験室に持ち帰り、環境DNA濃度を計測し、先に述べたモデルから湾内のマアジの個体数を推定しました。開発した手法の信頼性を評価するため、この野外調査では、計量魚群探知機を用いた音響計測(図3右)も同時に行い、2つの手法(環境DNA分析と音響計測)の間でマアジの個体数の推定値を比較しました。

写真提供:益田玲爾。
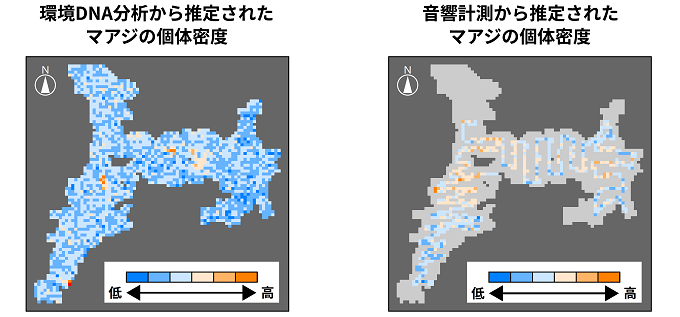
舞鶴湾内に生息するマアジの個体数は、音響計測を用いた手法では約1570万尾(信用区間(※2):1510万~1640万尾)、環境DNA分析を利用した手法では約3310万尾(信用区間:2320万~6320万尾)と推定されました。環境DNA分析による推定では、マアジの個体密度が極端に高い地点が見つかりました。この地点は舞鶴周辺で水揚げされた魚が多く集まる卸売市場のすぐ隣(湾の南西部)に位置していたことから(図4左)、市場から流入する大量のマアジのDNAを捉えたものと考えられます。そこで、この市場由来のDNAについて推定を補正したところ、湾内のマアジの個体数は約2230万尾(信用区間:770万~5290万尾)と推定されました。この値は、音響計測による推定値に比べて約42%大きく、精度も劣りますが、推定の誤差を踏まえれば2つの推定値は比較的良く一致しています。一方、マアジの個体密度の空間分布についても大まかな傾向が明らかになったものの、個々の地点の推定値は誤差が大きく、音響計測で推定された個体密度との関連も強くありませんでした(図4)。環境DNA分析によって個体数の空間分布を詳細に把握するためにはさらなる研究が必要と考えられます。
本研究によって、環境DNA分析を用いて海洋環境に生息する生物の個体数を推定できることが初めて実証されました。本研究の成功の鍵は、専門の異なる様々な研究者が研究分野の枠を越えて協働し、異分野の手法や技術を集約したことにあります。具体的には、調査船による舞鶴湾での野外調査とスキューバ潜水によるマアジ集団の観測(京都大 益田)、分子生物学実験による環境DNA濃度の計測(京都大 山本、村上、神戸大 源)、舞鶴湾の流動場解析と環境DNAトレーサーモデルの構築(北海道大 笠井、尹(現在は韓国・国立水産科学院)、計量魚群探知機を用いた魚類の個体数推定(北海道大 宮下、島根大 南)、環境DNAトレーサーモデルに基づく個体数の統計的推定法の構築(国立環境研 深谷、東北大 長田(現在は水産研究・教育機構))など、極めて多様な手法・技術が1つの研究の中で組み合わされています。環境DNA分析技術は、野外調査・実験室での分子生物学実験・モデリング等を有効に組み合わせることで、単なる対象生物の在・不在の判断を越えた、より広い応用的側面を持った技術に発展していくと期待されます。
今後の展望
水生生物の個体数を把握することは、水産資源の管理や希少種の保全を確実に行う上で不可欠です。しかし、水中で生活する生物を数えることはしばしば困難です。環境DNA分析は、目視や捕獲による従来の調査法の欠点を補い、水生生物の分布や量を効率的に把握するための有用な観測技術として注目を集めていますが、個体数推定の具体的な方法は確立していませんでした。本研究は、環境DNAの発生、移動、分解の過程を明確にすることによって、環境DNA濃度の多地点計測から水生生物の個体数を推定できることを示しました。この成果を嚆矢として、環境DNA分析による水域生態系の定量モニタリング技術の確立に向けた研究が加速することが期待されます。
注釈
※1流動場:対象の水域における水の流れ(速度)の分布を表したもの。
※2信用区間:推定値の精度を数値の区間で表現したもの。推定に用いた統計モデルの下で、推定対象の値を95%の確率で含むと考えられる区間を求めました。区間が短いほど推定の精度が高いと言えます。
研究助成
本研究は、JST 戦略的創造研究推進事業 CREST「環境DNA分析に基づく魚類群集の定量モニタリングと生態系評価手法の開発(研究代表者:近藤 倫生)」による支援を受けて行われました。
発表論文
【タイトル】
Estimating fish population abundance by integrating quantitative data on environmental DNA and hydrodynamic modeling
【著者】
Keiichi Fukaya, Hiroaki Murakami, Seokjin Yoon, Kenji Minami, Yutaka Osada, Satoshi Yamamoto, Reiji Masuda, Akihide Kasai, Kazushi Miyashita, Toshifumi Minamoto, Michio Kondoh
【雑誌】
Molecular Ecology
【DOI】
10.1111/mec.15530
【URL】
https://onlinelibrary.wiley.com/doi/10.1111/mec.15530【外部サイトに接続します】
問い合わせ先
【本研究の詳細に関する問い合わせ】
国立研究開発法人国立環境研究所 生物・生態系環境研究センター
生物多様性評価・予測研究室 特別研究員 深谷肇一
【研究プロジェクトに関する問い合わせ】
東北大学大学院生命科学研究科
教授 近藤倫生
【報道に関する問い合わせ】
国立研究開発法人国立環境研究所 企画部広報室
kouhou0(末尾に@nies.go.jpをつけてください)
029-850-2308
国立大学法人東北大学大学院 生命科学研究科広報室
lifsci-pr(末尾に@grp.tohoku.ac.jpをつけてください)
022-217-6193


