空間的に反復された環境DNAメタバーコーディングのための多種サイト占有モデリングと研究デザイン
論文情報
- タイトル
- Multispecies site occupancy modelling and study design for spatially replicated environmental DNA metabarcoding
空間的に反復された環境DNAメタバーコーディングのための多種サイト占有モデリングと研究デザイン - 著者
-
Fukaya K., Kondo N. I., Matsuzaki S. S., Kadoya T.
深谷 肇一、今藤 夏子、松崎 慎一郎、角谷 拓
- 雑誌
- Methods in Ecology and Evolution DOI: 10.1111/2041-210X.13732
- 受理・掲載
- 2021年9月7日 受理, 2021年10月4日 オンライン掲載

概要
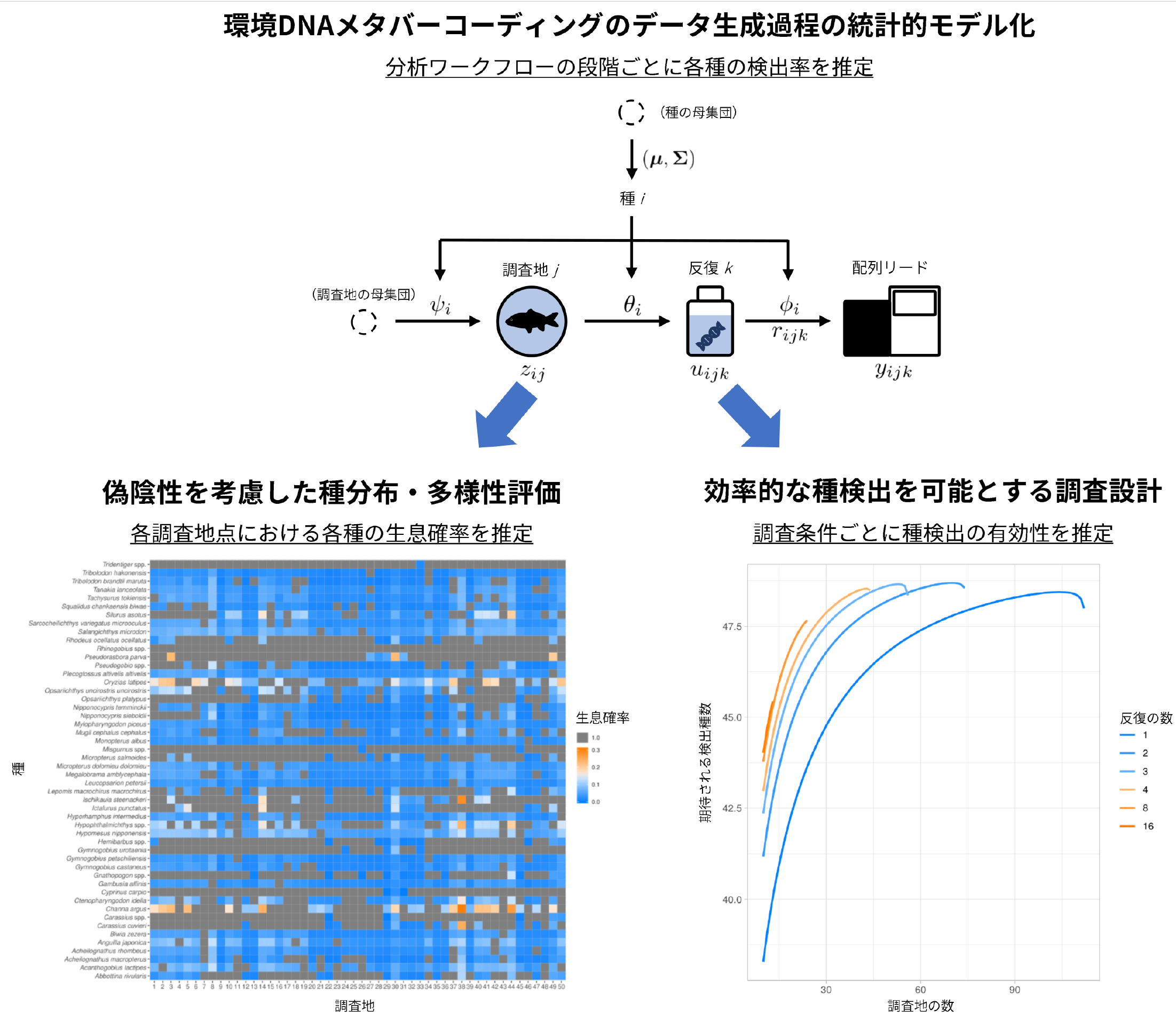
「環境DNAメタバーコーディング」は、水などの環境中に含まれるDNA分子を分析することで、そこに生息する特定の分類群(魚種など)を網羅的に検出する手法です。生息環境の破壊が少なく、コスト効率の高い生物多様性評価法として注目されています。しかし、環境DNAメタバーコーディングでは、本当は生息している種を検出できない誤差(偽陰性)が生じることも指摘されています。環境中のDNA分子の濃度は一般に非常に低く、サンプリングや分析の過程でDNA分子の捕捉や検出ができない場合があることが原因だと考えられます。本研究では、環境DNAメタバーコーディングで生じるこうした偽陰性を考慮して種の分布や多様性を評価するための新しい統計解析手法を提案しました。
この手法では、環境DNAメタバーコーディングによる種検出の過程を明示的に表す統計モデルを用いることで、分析ワークフローの異なる段階における偽陰性の生じやすさを定量し、検出されなかった種が実際には調査地点に生息している確率を評価します。また、偽陰性の生じやすさに関する情報に基づき、限られた予算の下で効率的に多くの種を検出できる調査デザインを特定できます。
提案手法を霞ヶ浦流域の淡水魚群集のデータに適用したところ、環境DNAメタバーコーディングによる種の検出率には大きな不均一性があり、特定の種が偏って検出されやすい特徴があることが分かりました。また、偽陰性を防いでより多くの種を検出するためには、調査地点ごとに環境試料の反復を複数取得することが好ましいことが示されました。
本研究で提案された枠組みを用いることで、環境DNAメタバーコーディングによる種多様性のモニタリングをより詳細かつ効率的に行えるようになると考えられます。今後は、本提案手法を容易に扱うことができるソフトウェア(Rパッケージ)の開発を進めていきます。