方法
DNAバーコーディングの情報収集
本プロジェクトでは、可能な限り標本の非破壊DNA抽出を行っています。
非破壊DNA抽出とは、標本の一部を外したり、すり潰したりしないでDNAを抽出する方法です。
DNAを抽出した後でも、同一の標本について形態と塩基配列情報を比較することができます。
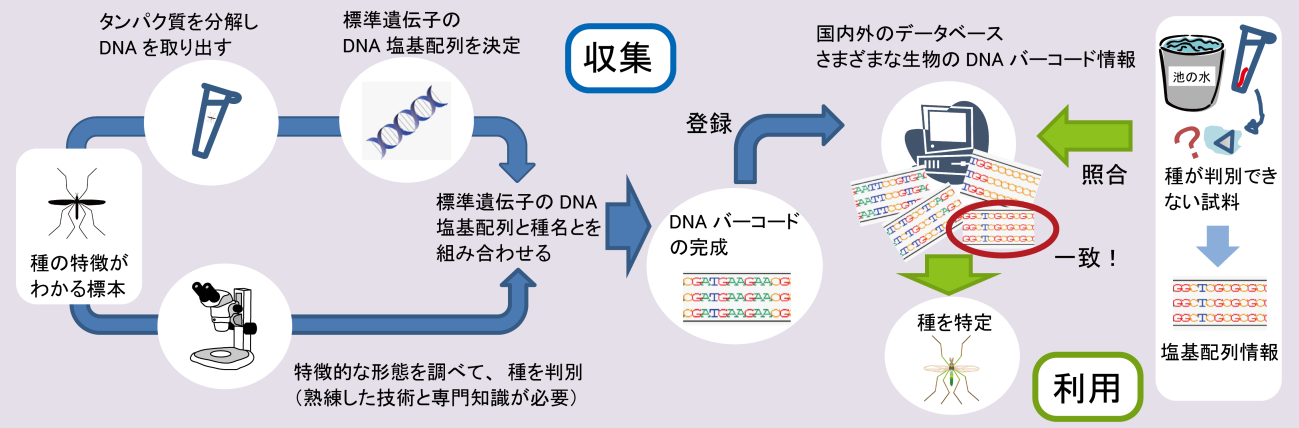
種の特徴がわかる標本のタンパク質を分解しDNAを取り出し、標準遺伝子のDNA塩基配列を決定する。また同標本の特徴的な形態を調べて種を判別する(熟練した技術と専門知識が必要)。DNA塩基配列と種名を組み合わせ完成したDNAバーコードを国内外のデータベースに登録する。種が判別できない試料については、塩基配列情報をデータベースで照合し、種を特定することができる。
非破壊DNA抽出の形態同定におけるメリット
- 解剖の手間が省けて標本の取り扱いがより簡単になります
通常の標本作製では、10%のKOH水溶液でキチンの骨格以外を溶かして、封入剤を用いたプレパラートを作ります
(「日本のユスリカ」 pp 20-23)。この場合、翅を体に付けたまま処理してしまうとほぼ確実に変形するため、解剖して翅だけ別に扱う手間が生じます。
一方、本研究で用いているDNA抽出液の場合は、翅を一緒に浸漬しても変形の程度は小さいため、解剖の手間が省けて標本の取扱いがより簡単になります。
※しかし、中型標本のDNA抽出に適当な時間では、大型の標本が溶け残ったり、小型の標本が溶けすぎたりすることがあります。そのため、KOH処理と同様に、標本の大きさに合わせて抽出時間を調節する必要があります。
- DNAを再抽出できる可能性があります
DNA抽出後のサンプルは、ホイヤー液やバルサムで封入せず、ホールスライドにグリセリンで封入して観察後、99.5%エタノールに保存すれば、DNAを再抽出できる可能性があります。
DNAの抽出から塩基配列情報の取得まで
- 採集標本の保存
1.5mlのチューブに標本を入れた後、エタノール(99.5%が望ましい)で満たして保存します。
- DNA抽出
タンパク質分解酵素(プロティナーゼK)とDNA抽出バッファーをサンプルに加えます。
56°Cで数~十数時間、標本をすり潰さずに静置し、上澄みをDNA抽出液として保存します。
残った標本は、99.5%エタノールで保存し、形態観察やDNA抽出に利用します。
- PCR(Polymerase Chain Reaction)
COI遺伝子の一部である約700bpの領域を、PCRにより増幅します。
COIは、動物のDNAバーコーディングにおける標準遺伝子であり、シトクロームcオキシダーゼ・サブユニットIというタンパク質をコードしています。
プライマー(Folmer et al. 1994):
LCO1490(5’-GGTCAACAAATCATAAAGATATTGG-3’)
HCO2198(5’-TAAACTTCAGGGTGACCAAAAAATCA-3')
未精製DNAを用いるため、不純物の多いサンプルに適したDNA合成酵素を使用します。
- シークエンス
精製したPCR産物について、シークエンシング反応を行います。
シークエンサーでダイレクトシークエンスを行い、塩基配列情報を得ます。
- 塩基配列の解析
プライマー領域の除去などの塩基配列決定を行います。
配列間の相同性解析や、分子系統学的な解析を行い、バーコードとして登録します。


